
MetaGenoPolis (photos © INRA) : (1) Robot de triage des clones dans MetaFun (2) Conservation des échantillons dans SAMBO (3) Automate de séquençage de MetaQuant (4) Un milliard de séquences sur une petite plaque…
Qu'est-ce que le microbiote intestinal ? Comment se constitue-t-il ?
Le microbiote intestinal (auparavant appelé "flore") est constitué par les bactéries qui peuplent nos intestins.
A la naissance, le tube digestif est pourtant stérile. Mais il est immédiatement colonisé par un grand nombre de micro-organismes. Après 24 heures seulement, "l'intestin de chaque individu contient déjà près de 100 000 milliards de bactéries", précise l'Inra (Institut national de la recherche agronomique).
Un chiffre qui se retrouve tout au long de la vie : chaque adulte a 100 000 milliards de bactéries dans ses intestins, ce qui représente une masse d'environ 500 grammes (et constitue la moitié de nos selles). A titre de comparaison, il y a également environ 100 000 milliards de cellules dans le corps.
Qu'est-ce que la métagénomique ? Combien y-a-t-il d'espèces bactériennes dans le microbiote ?
 Ces bactéries intestinales ont chacune un génome, qui peut être étudié, séquencé de manière globale (technologies de séquençage haut débit).
Ces bactéries intestinales ont chacune un génome, qui peut être étudié, séquencé de manière globale (technologies de séquençage haut débit).Le génome de l'ensemble des bactéries du microbiote est appelé métagénome et son étude, la métagénomique. La métagénomique peut également être utilisée pour analyser le contenu génétique d'autres environnements complexes, comme les océans, les sols, etc.).
Selon un séquençage du métagénome d'échantillons de selles collectés auprès de 124 européens en 2009, il y aurait entre 1000 et 1150 espèces bactériennes différentes dans le microbiote ; chaque individu en hébergerait environ 160 (Junjie Qin et coll., Nature, mars 2010).
Chaque personne possède des bactéries relativement proches en termes d'espèces, mais la composition exacte de son microbiote (espèces, proportions) est unique.
Le microbiote est résilient
La composition du microbiote d'un individu est remarquablement stable dans le temps. En cas de perturbation, par exemple par un traitement antibiotique, le microbiote revient progressivement à un état proche de son état antérieur (résilience). Comme si la composition du microbiote était adaptée précisément aux caractéristiques spécifiques (génétiques ?) de notre organisme.
Si l'on souhaite modifier le microbiote pour raisons thérapeutiques, il faut donc le faire de manière répétée, par exemple en administrant régulièrement des bactéries sélectionnées pour leurs propriétés ou en effectuant à plusieurs reprises une greffe fécale (cf. infra),
A quoi sert le microbiote ?
Le microbiote est aujourd'hui considéré comme un "véritable organe du corps humain qui joue un rôle primordial pour la santé et le bien-être de l'homme", souligne l'Inra.
Il est en particulier impliqué dans l'immunité naturelle, la protection contre les pathogènes et les infections (effet "barrière" au niveau intestinal), la dégradation des composés de l'alimentation (fermentation, métabolisme). En sus de la défense anti-infectieuse, le microbiote influe donc également sur divers états pathologiques, comme l'obésité, le diabète, les maladies inflammatoires chroniques intestinales (MICI), ou encore les allergies.
Cela ouvre des perspectives thérapeutiques, mais encore faut-il connaître les facteurs bactériens aggravants ou favorisants, et savoir comment rééquilibrer ou modifier le microbiote pour influer sur l'évolution de ces pathologies. C'est tout l'enjeu des recherches actuelles, dont celles désormais menées par MetaGenoPolis.
MetaGenoPolis, un dispositif destiné à mieux comprendre l'impact du microbiote et à révéler d'éventuelles possibilités thérapeutiques
 Coordonné et hébergé par l'Inra, MetaGenoPolis associe l'Institut hospitalo-Universitaire de Cardiométabolisme et nutrition (ICAN) et l'Université Catholique de Lyon.
Coordonné et hébergé par l'Inra, MetaGenoPolis associe l'Institut hospitalo-Universitaire de Cardiométabolisme et nutrition (ICAN) et l'Université Catholique de Lyon.L'objectif ? "Démontrer l'impact du microbiote intestinal humain sur la santé et dans les maladies non-infectieuses, déterminer les composantes clés de l'interaction entre microflore et cellules intestinales, développer des approches pour moduler les populations microbiennes…", résume l'Inra.
MétaGénoPolis devrait permettre de démontrer que l'on peut "protéger, restaurer, greffer le microbiote intestinal à des fins préventives ou thérapeutiques, dans le domaine médical et nutritionnel" (traitements microbiens personnalisés, recommandations nutritionnelles).
MétaGénoPolis pourrait aussi identifier de nouveaux biomarqueurs de risque de maladies afin de "développer des outils non-invasifs de diagnostic précoce et de pronostic très précis, par exemple la détection du risque de syndrome métabolique ou de cirrhose du foie".
Pour cela, il faut créer des banques d'échantillons, les séquencer, identifier les interactions, propriétés, mécanismes moléculaires.
MetaGenoPolis : 4 plateformes innovantes pour cribler et séquencer des milliers d'échantillons par an
Financé à hauteur de 19 millions d'euros par les investissements d'avenir, MetaGenoPolis rassemble 4 plateformes innovantes au service de la communauté médicale, scientifique et industrielle :
- Sambo vise à créer une biobanque nationale constituée de plus d'un million d'échantillons intestinaux humains, en mesure de répondre à la demande croissante issue d'essais cliniques et de grands suivis épidémiologiques et nutritionnels.
- MetaFun est une plateforme de métagénomique fonctionnelle qui repose sur un ensemble de technologies de criblage à haut débit. Cet outil permet d'identifier les molécules et mécanismes du dialogue entre bactéries intestinales et cellules humaines (biomarqueurs, voie thérapeutique).
- MetaQuant est une plateforme de métagénomique quantitative : elle met en œuvre le séquençage d'ADN à haut débit et développe des outils de bio-informatique spécifiques pour la quantification de l'abondance relative des gènes et espèces bactériennes dans les échantillons intestinaux humains. En comparant les différents profils métagénomiques entre individus, les chercheurs peuvent définir le microbiote intestinal "normal", explorer les associations entre les microbes intestinaux et les maladies, la réponse aux traitements médicaux ou à l'alimentation. Ils peuvent également proposer un suivi dans le temps de l'effet d'interventions (médicaments ou aliments).
- Soca détermine l'impact sociétal et éthique de la recherche en métagénomique humaine dans le domaine de la nutrition et de la médecine.
Concrètement, toujours selon l'Inra, MetaGenoPolis permettra dès 2015 l'analyse de 7 000 échantillons chaque année (MetaQuant) et le criblage de 200 000 clones métagénomiques (clones bactériens, généralement de l'espèce Escherichia coli, qui "portent de grands fragments de génomes issus du microbiome intestinal humain") par MetaFun. Sambo pourra dans le même temps héberger 10 000 échantillons par an et en traiter plus de 2 000 de façon totalement automatisée.
Pour davantage d'informations sur MetaGenoPolis, vous pouvez consulter le site dédié mgps.eu et/ou lire la plaquette de présentation du projet, téléchargeable en cliquant ici et visualisable ci-dessous :
Un exemple concret récent chez l'homme : les effets positifs d'un transfert de microbiote sur la rectocolite hémorragique de 10 patients (étude pilote)
Comment modifier profondément et rapidement le microbiote d'un patient ? En lui faisant un lavement complet et en lui administrant, via un lavement ou une sonde de coloscopie par exemple, des selles prélevées chez un individu ne présentant pas de MICI. C'est la greffe fécale (encore appelée transfert fécal, bactériothérapie fécale, transplantation fécale, ou encore transplantation de microbiote fécal).
Cette méthode a été testée récemment auprès de 10 enfants et jeunes adultes, âgés de 7 à 20 ans et présentant une RCH "modérée à sévère".
Résultat, après 5 greffes fécales étalés sur 7 jours, 7 patients ont présenté une amélioration symptomatique une semaine après. Mieux, 3 patients n'ont plus eu aucun symptôme de RCH 1 semaine après le transfert fécal ! Cette amélioration s'est maintenue pendant un mois pour 6 patients, sans effets indésirables notables (Kunde S et coll., Journal of Pediatric Gastroenterology & Nutrition, juin 2013).
Cette amélioration est peut-être due à l'effet positif d'une ou plusieurs bactérie(s), absentes ou insuffisantes en cas de RCH, comme pourraient le suggérer d'autres études.
L'Inra a découvert une bactérie potentiellement anti-inflammatoire dans le microbiote
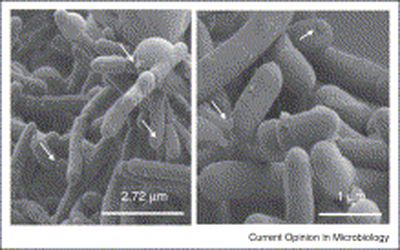 Gérard Corthier et d'autres chercheurs de l'Inra ont démontré en 2009 qu'une bactérie, Faecalibacterium prausnitzii, du genre Firmicutes (photo ci-contre), était moins présente dans le microbiote de patients atteints d'une rectocolite hémorragique (RCH) que dans le microbiote des personnes non atteintes (Soko H et coll., Inflammatory Bowel Diseases, août 2009). Ils ont donc formulé l'hypothèse que cette bactérie avait peut-être un rôle protecteur contre l'inflammation intestinale.
Gérard Corthier et d'autres chercheurs de l'Inra ont démontré en 2009 qu'une bactérie, Faecalibacterium prausnitzii, du genre Firmicutes (photo ci-contre), était moins présente dans le microbiote de patients atteints d'une rectocolite hémorragique (RCH) que dans le microbiote des personnes non atteintes (Soko H et coll., Inflammatory Bowel Diseases, août 2009). Ils ont donc formulé l'hypothèse que cette bactérie avait peut-être un rôle protecteur contre l'inflammation intestinale.Cette hypothèse a été vérifiée, pour le moment chez la souris, par Philippe Langella et son équipe, également de l'Inra : ils ont administré des souches bactériennes de Faecalibacterium prausnitzii à des souris, qui ont ensuite résisté à l'induction chimique d'une ulcération colique mimant une RCH (Miguel S et coll., Current opinion in microbiology, juillet 2013).
Peut-être que la réussite de la greffe fécale, évoquée au paragraphe précédent, était due au rajout de Faecalibacterium prausnitzii (provenant des donneurs sains) dans le microbiote des patients atteints de RCH ? Ou à celui d'autres bactéries ? De futures investigations devraient permettre d'en savoir plus.
En conclusion
Nous avons effleuré, via l'annonce de la création de MetaGenoPolis et le résultat d'études prometteuses, les enjeux de cette nouvelle science appelée métagénomique. Comme nous l'avons vu, l'affinement des connaissances sur les populations bactériennes intestinales pourrait permettre, à l'avenir, d'imaginer des traitements microbiens pour de nombreuses pathologies, en particulier en cas d'échec des traitements usuels.
Le transfert fécal, bien toléré mais un peu "lourd" à mettre en œuvre, pourrait être remplacé à moyen terme par l'administration de bactéries soigneusement sélectionnées pour leur rôle anti-inflammatoire ou immuno-modulateur.
Faecalibacterium prausnitzii et d'autres bactéries feront donc peut-être un jour partie de l'arsenal thérapeutique usuel !
Jean-Philippe Rivière
(Article édité le 12 août suite à la réception de remarques de Gérard Corthier : masse des bactéries ramenée à 0,5 kg, interprétation relativisée des explications possibles des résultats de l'étude sur les patients présentant une RCH).
En savoir plus :
Le site de MetaGenoPolis
"Une révolution, le microbiote au cœur du bien-être et de la santé humaine", communiqué de l'Inra, 2 juillet 2013
"A human gut microbial gene catalogue established by metagenomic sequencing", Junjie Qin et coll., Nature, mars 2010
"Investissements d'avenir : santé et biotechnologies", présentation sur le site gouvernement.fr
"Safety, Tolerability, and Clinical Response After Fecal Transplantation in Children and Young Adults With Ulcerative Colitis", Kunde S et coll., Kunde S et coll., Journal of Pediatric Gastroenterology & Nutrition, juin 2013
"Low counts of Faecalibacterium prausnitzii in colitis microbiota", Soko H et coll., Inflammatory Bowel Diseases, août 2009
- "Faecalibacterium prausnitzii and human intestinal health", Miguel S et coll., Current opinion in microbiology, juillet 2013
Ressources complémentaires (fichiers PDF téléchargeables en cliquant sur les liens) :
Plaquette du projet MétaGénoPolis (plaquette intégrée dans l'article ci-dessus)
Plaquette de présentation de Sambo, biobanque
Plaquette de présentation de Metafun, métagénomique fonctionnelle
Plaquette de présentation de Metaquant, métagénomique quantitative
Plaquette de présentation de Soca, éthique
Sources
Pour recevoir gratuitement toute l’actualité par mail Je m'abonne !

 7 minutes
7 minutes Ajouter un commentaire
Ajouter un commentaire


Commentaires
Cliquez ici pour revenir à l'accueil.